学院风采
学院新闻|我院路永钢老师课题组在蛋白质冷冻电镜投影图像方面提出三维重构新算法
学院新闻|我院路永钢老师课题组在蛋白质冷冻电镜投影图像方面提出三维重构新算法
蛋白质结构解析有什么用?
蛋白质结构解析是分子生物学的核心课题,对于人们认识蛋白质的功能,理解疾病的发病机理(例如病毒的致病机理),进行药物设计和疾病治疗等都具有非常重要的意义。近年来,冷冻电镜技术在测定生物大分子结构方面取得了突破性的进展,虽然目前DeepMind 公司开发的AlphaFold已经可以从蛋白质序列预测蛋白质的三维结构,但其准确性还有待提升,其结果也只能作为预测结果使用。
近日,必赢网址bwi437信息科学与工程学院路永钢课题组提出了一种基于球面嵌入的蛋白质三维重构算法,相关论文以“3D Reconstruction from Cryo-EM Projection Images Using Two Spherical Embeddings” (基于两次球面嵌入的冷冻电镜投影图像三维重构)为题在线发表于《Communications Biology》。路永钢教授课题组与必赢网址bwi437生命科学学院的朱莉副教授以及美国欧道明大学的He Jing教授合作完成了这项研究工作。该论文提出的算法有助于从冷冻电镜图像中重构出更加准确的蛋白质三维结构。
单颗粒分析(Single Particle Analysis)是冷冻电镜测定蛋白质结构的主流技术。在利用冷冻电镜获得大量同一种蛋白质分子的二维投影图像后,该技术利用三维重构算法可以计算出蛋白质的三维结构(如图1所示)。其中,蛋白质三维重构的核心问题是估计每个投影图像的投影方向,其本质是一个非凸优化问题。现有的算法大多是基于模板匹配,或者是基于期望最大化的参数估计算法,容易受到初始参数选取的影响,容易陷入局部极小,可能会重构出错误的蛋白质结构。
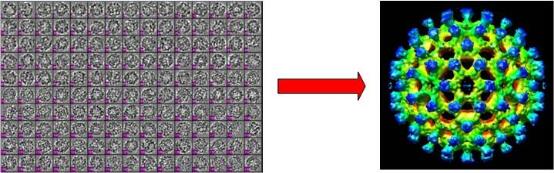
图1:从冷冻电镜的多个二维投影图像(左图)进行三维重构,获得蛋白质的三维结构(右图)(图片来自网络)
为了提升三维重构结果的可靠性,路永钢课题组在该研究工作中,充分利用了全体投影图像在投影方向以及等价线方面的全体一致性约束,通过两次球面嵌入获得了在三维空间中满足全体投影图像一致性约束的投影方向估计,进而计算出了蛋白质的三维结构。这种方法的特点是不需要初始模板,尽量从数据内部挖掘约束条件,对初始化依赖较小,因而提高了重构结果的可靠性和准确性。另外,路永钢课题组还提出了新的投影方向表示方法,利用两个互相垂直的向量(投影图像的法向量和自身坐标的X轴)来表示投影方向,并且讨论了这种表示和通常使用的欧拉角表示的等价性(见图2)
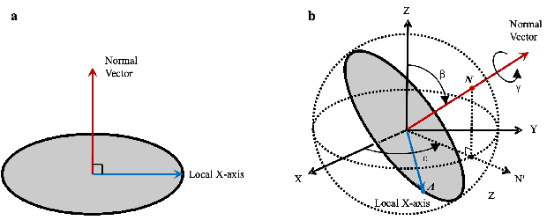
图2:投影图像的投影方向可以由其法向量和自身 X 轴向量表示,它们相互垂直。(a) 在投影图像自身坐标系中的两个表示向量;(b) 在三维重构空间中的两个表示向量,其中法向量可以用它在单位球面上的交点N来表示,自身 X轴向量可以用它在单位球面上的交点A来表示。欧拉角 b 是 Z 轴与法向量的夹角,欧拉角 a 是 X 轴与 N? 向量的夹角,N? 是法向量在 XY 平面上的投影,欧拉角 g 由自身 X 轴向量沿法向量的旋转角度确定。
在该论文的实验工作中,课题组分别使用了模拟数据集和两组真实数据集对算法进行了评价。通过与目前常见的几种算法(Synchronization、LUD、EMAN 2.1和RELION-2)进行对比,验证了所提算法的有效性。模拟数据由大肠杆菌70S核糖体对应的蛋白质结构通过计算机模拟投影生成。真实数据使用了从EMPIAR数据库下载的恶性疟原虫80S核糖体数据集(EMPIAR-10028)的冷冻电镜图像,以及Hedgehog受体补丁与纳米抗体TI23复合物(EMPIAR-10328)的冷冻电镜图像。实验结果证明了该论文提出的球面嵌入(Spherical Embedding)算法可以更准确地估计投影方向,并且在噪声比较高的情况下(例如SNR=0.1或0.2等),该算法能大大降低投影角估计的误差(见表1)。三维重构的结果也证明了利用该算法在不同噪声水平及不同数量的投影图像上进行重构时都具有一定的优越性,得到的重构结果具有更高的分辨率,也更加接近于真实结构(见图3)。
可见,利用该工作中提出的方法,可以有效改进冷冻电镜三维重构中的关键算法,即提高电镜图像投影方向估计的准确性,进而得到更加准确和分辨率更高的蛋白质三维结构。这为利用冷冻电镜获得更准确的生物大分子结构提供了一种有效的解决方案,对于冷冻电镜技术的发展和基于冷冻电镜技术的生物医学机理研究和应用研究都具有重要意义。
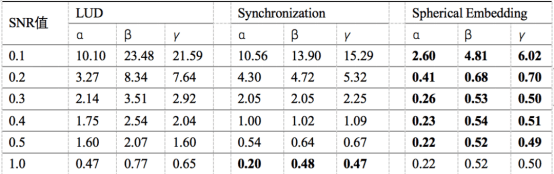
表1:对于含有不同噪声(SNR)的投影图像,不同算法估计的投影方向(欧拉角a、b、g)与真实结果之间的均方根误差(RMSE)比较。
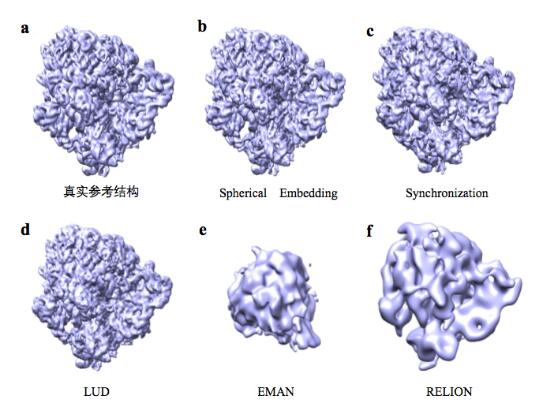
图3:从SNR=0.2的1000张模拟投影图像中重构出的蛋白质三维结构对比。a为真实结构(EMD-3508),b为利用该论文提出的球面嵌入算法得到的结构,c为利用Synchronization方法得到的结构,d为利用LUD方法得到的结构,e为利用EMAN 2.1得到的结构,f为利用RELION-2得到的结构。
论文下载地址:
https://www.nature.com/articles/s42003-022-03255-6.pdf
背景信息:
Communications Biology是Nature旗下于2018开始发刊的Open Access杂志(https://www.nature.com/commsbio/),主要发表生物科学领域的高质量研究和评论(该领域的重大进展或者新的生物学见解)。


